Next Generation Sequencing (NGS) och bioinformatik
LyssnaPå Folkhälsomyndighetens NGS-plattform utförs massiv parallellsekvensering av DNA och RNA. Plattformen används inom myndighetens övervakningsprogram och erbjuder även analyser inom myndighetens speciella diagnostik. Plattformen etablerades 2012 och används idag som primärt typningsverktyg för flera olika agens.
NGS-analyser
Flera olika NGS-baserade analyser erbjuds inom myndighetens speciella diagnostik. För sekvensering av enskilda smittämnen, se analyskatalogen. Beskrivning av mer generella sekvenseringsanalyser beskrivs nedan och de ingår även som referensdiagnostik i NRL nummer 37
Metagenomik för detektion av okänd patogen
För fall där man misstänker infektion men andra diagnostiska metoder inte kunnat påvisa patogen erbjuds metagenomisk sekvensering. Denna teknik innebär att all nukleinsyra i det kliniska provet sekvenseras och de resulterande data analyseras i detalj av erfarna bioinformatiker. Det genererade svaret presenteras som en omfattande teknisk rapport som inkluderar en detaljerad lista över databasträffar och en tolkning av potentiell klinisk relevans. Detta gör det möjligt att upptäcka och karaktärisera patogener med hög precision.
Metagenomisk sekvensering möjliggör en djupgående kartläggning av det genetiska materialet i det kliniska provet och ger därmed en bredare förståelse för den mikrobiologiska profilen. Svaret på metagenomisk sekvensering är resultatet av en samarbetsinsats mellan bioinformatiker och kliniska mikrobiologer, där den kliniska tolkningen bekräftar och kompletterar den tekniska analysen. För mer information om metagenomik för generell detektion av okänd patogen se:
16S metagenomisk sekvensering
16S-sekvensering utförs för att bestämma vilka bakterier som finns i ett kliniskt prov eller isolat. Till skillnad från metagenomisk detektion av okänd patogen, som sekvenserar allt tillgängligt genetiskt material, är 16S-sekvensering riktad mot ribosomens lilla subenhet och möjliggör identifiering och klassificering av bakterier med hög precision. 16S rRNA-genen är högt konserverat med vissa variabla regioner. Sekvensvariationerna mellan olika bakterier ger en unik identifiering, och analysen av dessa sekvenser möjliggör taxonomisk placering. Metoden är baserad på att amplifiera och sekvensera fyra variabla regioner av 16S rRNA-genen med två amplikon. Dessa amplikon omfattar V1V2 med storlek ca 350 bp och V3V4 460 bp. Dessa amplikon analyseras separat och klarar av tack vare stort sekvenseringsdjup att identifiera flera olika bakterier i samma prov. Metoden använder en intern kontrollsekvens (spike) som amplifieras och sekvenseras samtidigt med provet för att underlätta bedömning av halten av bakteriefynd samt att upptäcka amplifieringshämmande faktorer i provet som kan ge upphov till negativt resultat. Svaret består av två separata analyser som bl a summeras i en tabell med databasträffar och andelen av spik.
För mer information om 16S sekvensering se:
Helgenomsekvensering av bakterieisolat
För smittspårning eller frågeställningar kring antibiotikaresistens erbjuds helgenomsekvensering av bakterieisolat. Genom att sekvensera hela genomet hos bakterier får man en djupgående förståelse för deras genetiska sammansättning. Inom epidemiologisk smittspårning möjliggör denna teknik kartläggning av infektionsspridning och identifiering av smittkällor genom jämförelse av genetiska sekvenser. Denna analysen av bakteriers genetiska sammansättning kan också ge insikter om de specifika gener eller mutationer som kan vara kopplade till antibiotikaresistens.
För mer information om typning av olika bakterier se:
NGS bioinformatik
Bioinformatikerna vid Folkhälsomyndigheten har en central roll inom Avdelningen för Mikrobiologi genom att analysera omfattande biologisk data och utveckla sofistikerade mjukvaror. Deras fokus ligger på att skapa så kallade pipelines för att förbättra och automatisera analyser. Pipelines är seriekopplade dataprocesser som använder sig av mjukvaruverktyg för att strukturera, bearbeta och tolka genetisk information.
GENSAM
GENSAM (Gemensam nationell hantering av sekvenseringsdata inom klinisk mikrobiologi) är Folkhälsomyndighetens plattform och databas för automatiserad analys av helgenomsekvenseringsdata. Landets kliniska mikrobiologiska laboratorier som utför sekvenseringen lokalt deponerar genererade data som sedan analyseras centralt och delas med smittskyddsenheter samt berörda aktörer. I dagsläget hanteras SARS-CoV-2 av GENSAM men även andra smittämnen är under implementation. GENSAM har delfinansierats med hjälp av Vinnova och Europeiska kommissionen.
För frågor kring GENSAM skicka e-post till gensam@folkhalsomyndigheten.se.
Figur 1. Från prov till data till information.
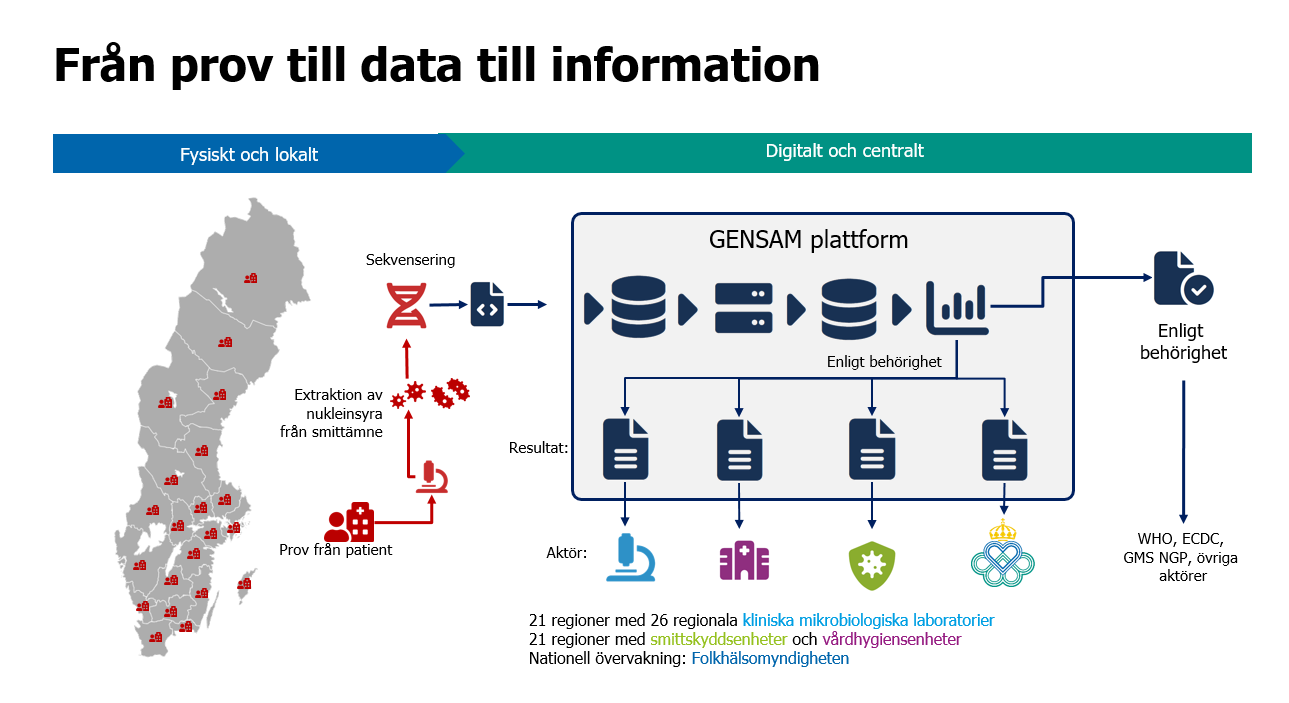
Patientprov förvandlas från data till information genom nukleinsyraextraktion och sekvensering av smittämnen. GENSAM-plattformen analyserar sekvensdata och delar resultaten med berörda aktörer. Berörda aktörer är 21 regioner med 26 regionala kliniska mikrobiologiska laboratorier, 21 regioner med smittskyddsenheter och vårdhygiensenheter samt Nationell övervakning: Folkhälsomyndigheten.

Kurser inom NGS och bioinformatik
Folkhälsomyndigheten erbjuder kurser inom NGS och bioinformatik. Dessa kurser är riktade mot laboratorier inom klinisk mikrobiologi som vill implementera eller håller på att implementera NGS-tekniken i sin verksamhet.
Har du frågor om kurserna? Kontakta till mikrobiologens kundtjänst på kundtjanst.mikrobiologen@folkhalsomyndigheten.se
